В конце двадцатого века на основе теории графов сформировалась новая область статистической физики – теория сложных сетей, ставшая эффективным инструментом исследования сложных систем различной природы, в том числе биологии и медицины [1]. В последние годы приложения теории сложных сетей к проблемам возникновения болезней человека привело к возникновению нового направления в медицине – сетевой медицины (Network Medicine) [2-4]. Цель данной статьи – представить краткий обзор наиболее важных публикаций этого научного направления, прежде всего связанных с пониманием проблемы взаимосвязи различных заболеваний.
В современной биологии и медицине значительные усилия направлены на нахождение связей между молекулярно-генетическим происхождением заболевания и его фенотипическим проявлением в виде симптомов. Хотя часто заболевания лечатся независимо от других, мало кто сомневается, что болезни связаны между собой. В 2007 году была построена первая сетевая структура заболеваний человека, в которой каждому узлу соответствует определенное заболевание и между узлами существует связь, если соответствующие им заболевания вызваны каким-то одним генетическим изменением [2]. Позднее аналогичные сетевые структуры стали создавать на основе патологий в метаболических реакциях, в белковых взаимодействиях, регуляторных и сигнальных сетях и т.д., а также учитывались взаимодействия людей в социальных сетях, что позволило начать изучение проблем медицины методами теории многослойных сетей [3].
Чаще всего болезнь вызвана взаимодействием процессов на молекулярном уровне. Взаимосвязи между этими процессами отображаются в интерактоме – многослойной сетевой структуре включающей в себя все взаимодействия в живой клетке: от сети взаимодействий белок-белок до регуляторных сетей и сетей метаболических реакций. Несколько лет назад стало известно свойство связанных с болезнями белков взаимодействовать между собой. Это дало основание предположить, что такие белки образуют в интерактоме кластер, содержащий все связанные между собой молекулярные субстраты болезни. Этот кластер получил название модуль заболевания.
Сети заболеваний человека
Современная система классификации заболеваний человека была основана в конце девятнадцатого века из наблюдений корреляций между анализом патологий и клиническими синдромами и в значительной степени зависела от искусства определения синдромного фенотипа. В течение двадцатого века эта процедура стала более объективной, поскольку стала опираться еще и на молекулярно-биологические диагностические тесты. Тем не менее, эта классическая диагностическая схема имеет недостатки, признаваемые многими специалистами как из-за отсутствия точности в выявлении доклинической стадии заболевания, так из-за отсутствия специфичности при попытках определить заболевание однозначно. Однако, последние успехи молекулярной биологии дают возможность современной медицине диагностировать заболевания гораздо более точно.
В качестве примера можно привести недавние исследования, которые показали, что если у человека одна из аллелей содержит ген FTO, то у этого человека риск ожирения возрастает на 30% , а если этот ген содержится в обеих аллелях, риск ожирения возрастает до 67%. Такая связь между геном FTO и ожирением может служить примером сильной прямой связи между генотипом и фенотипом. При этом избыточный вес оказывается тесно связанным такими заболеваниями, как сахарный диабет, астма и другими.
Недавно Кристакис и Фаулер изучив данные многолетних медицинских наблюдений различных социальных групп в одном из районов штата Массачусетс (США), обнаружили, что социальные связи различной природы оказывают на возникновение избыточного веса не меньшее влияние, чем генетические факторы. Например, вероятность того, что если друг вашего друга достаточно хорошо знаком с человеком с избыточным весом, он сам наберет избыточный вес, на 20% выше, чем в социальных сетях со случайными связями между людьми. Было показано, что ожирение подобно инфекциям распространяется по социальным сетям.
В настоящее время имеется множество данных, в которых устанавливаются связи между заболеваниями в виде сетей. Например, Rzhetsky et al установили связи между 161 сопутствующими друг другу заболеваниями на основе данных взятых из истории болезней полутора миллиона пациентов по программе Medicare [5]. Hidalgo et al построили сеть фенотипических признаков заболеваний используя данные о сопутствующих друг другу болезнях более 30 миллионов пациентов [6], показав, симптомы не только по-прежнему являются решающим фактором правильного клинического диагноза и успешного лечения, но и важнейшим ресурсом для теоретического анализа.
Выявление заболеваний из неполной интерактомы
Несомненно, важно и необходимо уметь выявлять мутации, ведущие к заболеваниям, в контексте интерактома человека – сетевая структура, всесторонне отражающей все биологически значимые молекулярные взаимодействия. Однако, предсказательные возможности современных подходов, основанных на теории сложных сетей, ограничены множеством причин. Во-первых, сегодня нам известно лишь около 20% общей структуры интерактома и поэтому в поисках молекулярных механизмов заболеваний вынуждены опираться на пока еще очень неполные данные о сети белковых взаимодействий. Во-вторых, как уже упоминалось выше, продукты мутаций генов – соответствующие белки – рассеяны по интерактома не случайным образом, а имеют тенденцию взаимодействовать друг с другом, формируя один из нескольких подграфов, называемым модулем заболевания (disease module).
В работе [8] даны примеры взаимодействий модулей заболеваний для рассеянного склероза (multiple sclerosis (MS)), пароксизмальные расстройства (peroxisomal disorders(PD)) и ревматоидного артрита (rheumatoid arthritis (RA)). Заболевания с перекрывающимися модулями (MS и RA) имеют фенотипические сходства и высокую вероятность быть сопутствующими заболеваниями. Не пересекающиеся модули заболеваний, такие как MS и PD, не имеют выявленных общих клинических симптомов.
Однако, несмотря на частое использование или просто упоминания в научных дискуссиях и докладах, гипотеза о модулях заболевания не имеет конкретных алгоритмов и правил их выявления и построения, а также сколько-нибудь надежного обоснования с точки зрения современной теории сложных сетей.
Фрагментация модулей заболеваний
Построение интерактома было начато с составления сети из 141296 физических взаимодействий между 13460 белками , экспериментально задокументированных в клетках человека. Сюда вошли белок-белковые, регуляторные взаимодействия и метаболические реакции. Также был составлен список 299 заболеваний из медицинского рубрикатора заболеваний (Medical Subject Headings (MeSH)) содержащий не меньше 20 связанных генов на основе данных, содержащихся вMendelian Inheritance in Man (OMIM) и баз данных genome-wide association study (GWAS), включающих в себя 2436 белков, связанных с болезнями. Несмотря на некоторый прогресс, значительна часть интерактома из списка болезней, связанных с заболеваниями генов остается несвязанной.
Например, для рассеянного склероза из 69 белков всего 11 образуют связанный подграф, остальные же случайным образом рассеяны по интерактому.
Эта картина имеет место для всех 299 заболеваний. Их выявленные модули составляют около 20% соответствующих генов болезни.
Существует несколько причин фрагментации интерактома как сетевой структуры. Основной причиной является недостаточность данных: отсутствующие связи оставляют многие связанные с заболеваниями белки изолированными в их модулях заболеваний. Согласно важнейшему разделу теории сложных сетей — теории перколяции, если имеется определенная доля p связей, связанный подграф (в данном случае модуль заболевания) содержащий m узлов, при значения р выше критического значения испытывает фазовый переход при котором формирует наблюдаемый модуль. Если же это значение ниже критического, модуль остается слишком фрагментированным и не воспринимается как нечто целое.
Детальные вычисления показали, что согласно теории перколяции, для заболевания причиной которого было меньше 25 генов, его модуль слишком фрагментирован, чтобы быть наблюдаемым в интерактоме. То есть, только заболевания, причиной которых было больше 25 генов, имеют хорошо наблюдаемые модули заболеваний (Рисунок 1).
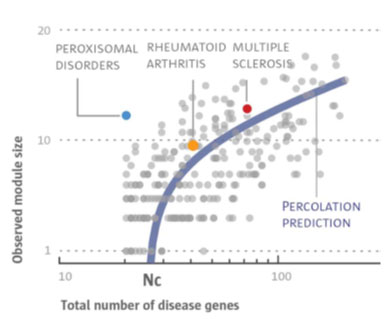
,Рисунок 1. Оценка критического размера модуля заболевания на основе теории перколяции
Фенотипическая Сеть Болезней (Phenotypic Disease Network (PDN))
Хотя успехи в использовании сетевых методов на молекулярно-генетическом уровне для изучения различных заболеваний весьма впечатляющи, немало полезной информации можно получить применяя сетевые технологии при анализе фактических данных по заболеваниям на фенотипическом уровне в виде истории болезней пациентов, содержащих информацию о связях различных заболеваний между собой и прогрессе этих заболеваний.
Чтобы понять связи болезней, каждую из них определяют как определенный специфический набор фенотипов влияющих на одну или несколько функциональных систем. При этом подразумевается, что живой организм может быть описана в сложном фенотипическом пространстве как связанная из отдельных элементов дискретная система. Для этого в 2009 году была построена Фенотипическая Сеть Болезней (Phenotypic Disease Network (PDN)) на основе изучения корреляций историй болезней более тридцати миллионов пациентов [6] .
Было установлено, что болезни прогрессируют и развиваются преимущественно вдоль связей этой сети и, более того, было выявлено, что такое развитие различно для пациентов разного пола и различных рас. Было установлено также, что пациенты, страдающие заболеванием, имеющим большое числом связей, умирают раньше, чем пациенты с заболеванием с небольшим числом связей. В этом исследовании была создана онлайновая запрашиваемая база данных из 18 различных наборов данных, полученных из истории болезней от более 31 миллиона пациентов. Связи (сообщества) заболеваний могут быть изучены в режиме онлайн или загружены в полном объеме.
Обозначим ![]() - (Relative Risk of disease pairs i and j) относительный риск заболеть двумя заболеваниями для одного и того же пациента
- (Relative Risk of disease pairs i and j) относительный риск заболеть двумя заболеваниями для одного и того же пациента
где Cij — число пациентов, страдающих обоими заболеваниями, N — полное число пациентов в популяции Pi и Pj число пациентов с заболеваниями соответственно i и j, то есть распространенность заболеваний i и j
Корреляция Пирсона Фij как мера между двумя бинарными переменными
![]()
Эти две меры различия между сопутствующими заболеваниями (comorbidity) не являются абсолютно независимыми. Например, они обе растут с ростом числа пациентов, страдающих обоими заболеваниями.
При этом сеть, построенная на основе RR связей, переоценивает расстояния между редкими заболеваниям, в то время как Ф недооценивает расстояния между парами редких заболеваний.
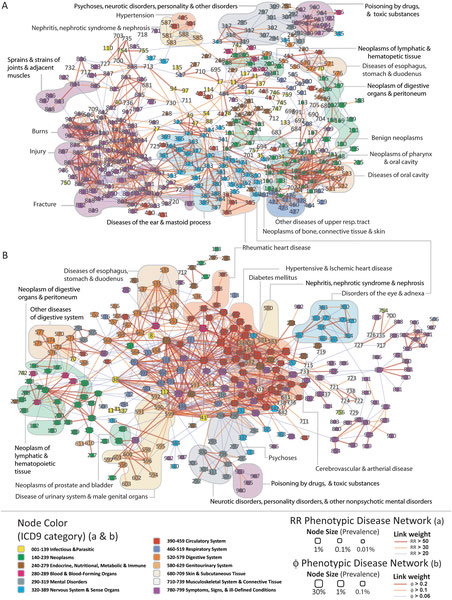
Рисунок 2. ( A) Фенотипическая Сеть Болезней (Phenotypic Disease Network (PDN)) , построенная по RR связям, образует своеобразное пространство фенотипов заболеваний (B) . Фенотипическая Сеть Болезней (PDN) , построенная по Ф связям
Основные результаты
В сети PDN узлами являются фенотипы заболеваний, описываемые уникальными ICD9 кодами, состоящим из 5 цифр. Первые три цифры обозначают основную категорию заболевания, а оставшиеся две цифры дают дополнительную информацию о заболевании. В совокупности, ICD9 классификация состоит из 657 различных категорий на уровне 3 цифр и 16459 категорий на уровне 5 цифр.
Используя только наиболее сильны и важные связи между узлами в сети PDN, предлагается два упрощенных варианта этой сети: первый на основе RR связей (Рисунок 1A) и второй на основе карреляционных Ф связей (Рисунок 1B).
У этих двух версий есть некоторые сходства, например, в обоих выявляется связь между гипертонией и почечными заболеваниями. При этом в первой версии мы видим сравнительно редко встречающиеся заболевания и в ней присутствуют четко различающиеся сообщества (модули), в то время как во втором варианте PDN сети больше широко распространенных заболеваний и связей между разными категориями ICD9 кода.
Динамические свойства фенотипической сети заболеваний
PDN сеть представляет собой своеобразную «карту» заболеваний, по которой можно изучать их развитие. Это развитие можно рассматривать как динамический сетевой процесс, когда пациенты «перепрыгивают» от одного заболевания к другому вдоль связей PDN сети.
Основываясь на имеющихся статистических данных о влиянии половых и расовых различий на RR связи PDN сети
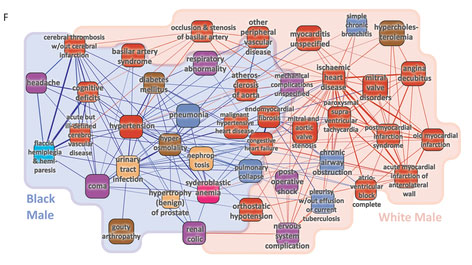 Рисунок 3. Различие в связях между заболеваниями в сети PDN в зависимости от этнического и полового различия на примере белых и афроамериканских мужчин
Рисунок 3. Различие в связях между заболеваниями в сети PDN в зависимости от этнического и полового различия на примере белых и афроамериканских мужчин
В заключение, обсудим вопрос о связи смертности от заболевания i в зависимости от числа его связей с другими заболеваниями. Количественно число связей данного заболевания i вычисляется стандартными формулами теории сетей ![]() и
и ![]()
Идеи и методы этого исследования были развиты для построения сети метастаз и создания модели развития ракового заболевания [10].
Сети белковых взаимодействий при сахарном диабете второго типа
В статье [7] на остове многочисленных биологических баз данных построены сетевые структуры белок-белковых взаимодействий для клеток поджелудочной железы при сахарном диабете второго типа, проанализированы свойства состояний в норме и патологии и сделаны сравнивая со свойствами случайных сетей Эрдеша-Реньи, сетей «тесного мира» Уоттса-Строгатца, а также сетей, построенный по конфигурационной модели, в которых сохраняется последовательность степеней узлов, следовательно, сохраняется и степенной закон сетей (scale free networks).
Распределение числа узлов по числу связей в сетях белок-белковых взаимодействий в клетках поджелудочной железы в норме и патологии является степенным. Такие сети со степенным законом распределения узлов по числу связей получили название безмасштабных сетей.
Таблица 1. Основные характеристики двух сетевых структур белок-белковых взаимодействий в норме (N1 и N2) и патологии (D1 и D2) при заболевании сахарным диабетом второго типа. N — общее число узлов (белков) в сети, NC - общее число связей в сети, ‹ k › — среднее число связей узла сети, D — диаметр сети, ‹CC› - среднее значение кластеризации сети.
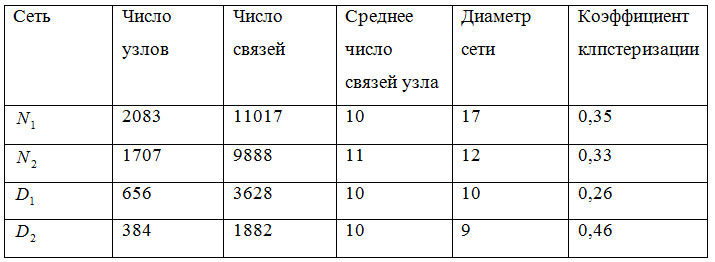
Анализ свойств сетей белковых взаимодействий в норме и патологии показывает, что сети здоровых клеток поджелудочной железы являются более случайными, чем сети больных клеток и такое отсутствие достаточно высокой случайности сети, по –видимому и ведет к заболеванию.
Для подтверждения этой гипотезы целесообразно вычислить энтропию сетей белковых взаимодействий здоровых и больных клеток.
Заключение
Описанные выше примеры далеко не исчерпывают основные направления приложения сетевых методов в медицине и биологии. Например, можно упомянуть статью, где описывается создание сетевой структуры “Лекарство — Белок (Drug – Turget)” [11]. Можно констатировать, что появившиеся в последние годы инструменты сетевой медицины на основе методов теории сложных сетей и современных баз данных взаимодействий биологических компонентов живой клетки на молекулярно-генетического уровня формируют основу для систематического изучения не только сложных молекулярных механизмов конкретных заболеваний. Прогресс в этом направлении имеет важное значение и для выявления новых генетических причин болезней и ассоциированных с заболеваниями мутаций негенетического происхождения, а также создает предпосылки для более эффективного поиска лекарств и биомаркеров для сложных заболеваний.
Литература
- Barabasi A.-L. Network Science. http://barabasi.com/networksciencebook/
- Goh et al. Human disease network. PNAS. May 22, 2007, vol. 104, no. 21, 8687
- Barabasi et al. Network medicine: a network based approach to human disease. Nature Review. Genetics. vol.12, January 2011, 57
- Zhou X. Z., Mencht J., Barabasi A.L., Sharma A.. Human symptoms-disease network. Nature Communications. 2014, 5-4212.DOI: 10.1038
- Rzhetsky, A., Wajngurt, D., Park, N & Zheng, T. Probing genetic overlap among complex human phenotypes. PNAS. 104, 11694-11699 (2007).
- Hidalgo C. A., Blumm, N., Barabasi A.L.& Christakis, N. A dynamic network approach for the study of human phenotypes. PLos. Comput.Biol. 5, e1000353 (2009)
- Rai, A., Pawar. K.A., and Jalan, S. Prognostic interaction patterns in diabetes mellitus II: A random-matrix-theory relation. Physical Review E 92, 022806 (2015)
- Menche, J., Sharma, A., Kitsak, M., Ghiassian, S. D., Vidal, M., Loscalzo, J., Barabási, AL. Uncovering disease-disease relationships through the incomplete interactome. Science. 2015 • v. 347 ISSUE 6224, pp.841-848
- Ghiassian, S. D., Menche, J., Barabási, A.-L. A DIseAse MOdule Detection (DIAMOnD) in Algorithm Derived from a Systematic Analysis of Connectivity Patterns of Disease Proteins in the Human Interactome. PLOS Computational Biology DOI:10.1371. April 8, 2015
- Chen LL Blumm N, Barabasi A-L, Christakis NA and Deisboeck (2009). Cancer metastasis networks and the prediction of progression patterns. British Journal of Cancer (2009) 101, 749–758.
- Yildrim M., Goh K., Gusick M., Barabasi AL., & Vidal M. Drug-target network. Nature Biotechnology. 25, N 10. Oktober 2007.
